一、理论知识
1.1 K-Means
给定一组数据集,聚类算法将它们分成不同的子组。我们希望类内实例高度相似,类间实例低相似。
在样本集中,随机选取K个点作为中心$\bold \mu_k$,计算每个样本到中心点的距离,并将样本划分到离它最近的那个点的集群中。使用变量$r_{nk}$表示数据样本$\bold x^{(n)}$是否属于集群k: $$ r_{nk}=\left{\begin{matrix}1,k=arg\min_j||\bold x^{(n)}-\mu_j||^2\0,otherwise\end{matrix}\right. $$ 对于每个集群,用所有样本的平均位置更新中心点的位置: $$ \mu_k=\frac{\sum^N_{n=1}r_{nk}\bold x_n}{\sum^N_{n=1}r_{nk}} $$ 重复上面的样本分配和中心更新过程即可,该过程是保证收敛的。类内距离之和会随着K的增大而减小,因此选择K的时候考虑寻找使得距离基本收敛的K值或是依据下游应用需求选择K值。
K平均聚类的结果高度依赖于初始化的中心点。选择初始化中心点的一般方法有:
- 随机选择:随机选择数据样本作为初始中心。问题在于有可能选择了距离很近的多个样本作为初始中心
- 基于距离的方法:一开始随机选一个样本作为中心点,其他时候选择离各个中心最远的样本作为中心点。问题在于可能选到异常值
- 随机+距离方法:第一个中心点为随机选择的样本,从全部远离现有中心的样本中随机选择下一个中心
一个点要么属于一个集群,要么不属于,存在赋值难的问题。可以用软K平均算法解决: $$ r_{nk}=\frac{e^{-\beta}||\bold x^{(n)}-\mu_k||^2}{\sum^K_{i=1}e^{-\beta}||\bold x^{(n)}-\mu_i||^2} $$ 这样一来,$r_{nk}$可以理解为样本属于某个集群的几率。
该算法还对异常值敏感。而且欧几里得距离决定了决策边界是球形的,当不同簇的形状不规则时,性能可能不佳。
1.2 GMM与EM算法
在监督学习中,回归和分类都可以理解为学习的条件概率分布。类似地,从概率建模的角度来看,无监督学习可以理解为对输入数据的概率分布进行学习,最简单的方法是假设该分布为高斯形式。但是一般情况下,样本的分布比高斯分布复杂得多。为了更好地建模,就需要更好的分布表示方法,即使用复杂的分布,但是训练成本又会增加。
因此,一个直观的思路是使用很多简单模型的加和表示复杂模型: $$ p(\bold x)=\pi_1p_1(\bold x)+\pi_Kp_K(\bold x) $$ 其中,所有$\pi$的和为1,用来使得分布合法。而每个$p_i$表示简单的分布。上面的每一项又可以看作是联合分布的边缘分布: $$ p_z(\bold x)\pi_z=p(\bold x|z)p(z)=p(\bold x,z) $$ 这种模型被称为潜变量模型LVM。我们将各个分布看做是高斯分布,则该模型又称为高斯混合模型,即GMM。如果一个高斯混合模型由$K$个高斯分布组成,则总的分布表达式为: $$ p(\bold x)=\sum^K_{k=1}\pi_kN(\bold x;\bold\mu_k,\bold\Sigma_k) $$ 其中,$K$表示高斯分布的个数,$\pi_k$表示第$k$个分布的权重。$\sum^K_{k=1}\pi_k=1$。$\bold \mu_k$和$\bold \Sigma_k$是第$k$个高斯分布的均值向量和协方差矩阵。
EM算法可以学习高斯混合模型的参数,使得该模型在特定的数据集下获得更大的对数似然函数值。算法分为E和M两步。E指评估预期,M指更新数据:
- E-step:$Q(\bold\theta;\bold\theta^{(t)})=E_{p(\bold z|\bold x;\bold\theta^{(t)})}[\log p(\bold x,\bold z;\bold \theta)]$
- M-step:$\bold\theta^{(t+1)}=\arg\max_\bold\theta Q(\bold \theta;\bold\theta^{(t)})$
对于上述的高斯混合模型,在E-step中,可以求得对数似然函数为: $$ \log p(\bold x,\bold z;\bold \theta)=\sum^K_{k=1}z_k\cdot[\log N(\bold x_n|\bold \mu_k,\bold\Sigma_k)+\log\pi_k] $$ 令: $$ \gamma_k^{(t)}\triangleq\frac{N(\bold x_n|\bold \mu^{(t)} k,\bold\Sigma^{(t)}_k)\pi_k}{\sum {i=1}^KN(\bold x_n|\bold \mu^{(t)} k,\bold\Sigma^{(t)}_k)\pi_i} $$ 则有: $$ Q(\bold\theta;\bold\theta^{(t)})=\sum {k=1}^K\gamma_k^{(t)}[\log N(\bold x_n|\bold \mu_k,\bold\Sigma_k)+\log\pi_k] $$ 然后在M-step更新各个参数: $$ \bold\mu_k^{(t+1)}=\frac1{N_k}\sum^N_{n=1}\gamma_{nk}\bold x_n\ \bold\Sigma_l^{(t+1)}=\frac1{N_k}\sum^N_{n=1}\gamma_{nk}(\bold x_n-\mu_n^{(t+1)})(\bold x_n-\mu_n^{(t+1)})^T \pi_k^{(t+1)}=\frac{N_k}N $$ 其中$N_k=\sum^N_{n=1}\gamma_{nk}$,表示第$k$类的样本的有效数量。反复执行E和M两步训练参数,直至模型收敛或到达最大训练次数即可。
二、代码实现
2.1 K-means
在K-means算法中,我们将训练数据集分成若干个聚类,对每个类打上标签后对训练样本进行分类,将属于的类的标签当做预测结果即可。
2.1.1 中心点的选择
K平均算法的核心就是中心点的选择和更新。对于本次实验使用的MNIST数据集,样本共有10类,因此聚类时选择10个不同的中心点。初始化中心点有多种方法,我会在之后的实验结果分析中分析不同的初始化方法对结果的影响。
下面的代码以随机选择为例子。在全部样本点中选择十个样本点作为初始化中心:
```python
设置基本参数
k = 10 # 类别数 t = 100 # 迭代次数
生成初始中心
完全随机
center_points = np.random.choice(train_images.shape[0], k, replace = False).tolist() for i in range(len(center_points)): center_points[i] = train_images[center_points[i]] center_points = np.asarray(center_points) ```
同时,我还对基于距离的方法进行了尝试。即,第一个中心点为随机选择的样本点,第二个中心点为离第一个中心点最远的样本点,第三个中心点为距离第一和第二个中心点距离之和最大的样本点,以此类推:
python
center_points = genCenterPoints(train_images, k)
```python
基于距离选择中心点
def genCenterPoints(train_images, k): # 第一个中心点随机选择 center_points = [train_images[random.randint(0,train_images.shape[0]-1)].tolist()] while len(center_points) != k: print(len(center_points)) dist = None point = None # 遍历每个样本点 for each in train_images: # 不考虑已经作为中心点的样本点 if each.tolist() in center_points: continue # 计算当前样本到各个中心点的距离之和 tmp_dist = np.sum(np.linalg.norm(np.asarray(center_points) - each,axis = 1)) # 选择距离最短的样本点作为下一个中心点 if dist==None or tmp_dist<dist: point = each dist = tmp_dist center_points.append(point.tolist()) return np.asarray(center_points) ```
2.1.2 训练过程
之后就是对各个样本点进行分类和中心点的迭代更新:
```python
进行迭代
for i in range(t): # 对训练样本进行分类 r = classification(center_points,train_images, k) # 更新中心点位置 center_points = updateCenterPoints(r, train_images, k) ```
对样本点进行分类时,使用数组
r[i][j]
表示第
i
个样本是否属于第
j
类。是则为1,否则为0。计算出当前样本和各个中心点之间的距离,选择最近的中心点作为该样本属于的类别即可。这里我调用了
np.linalg.norm
来计算距离,得到的是欧式距离,也就是各个特征值的平方和开根号作为距离。样本距离可以采用不同的标准,会对分类的结果产生不同的影响。
python
def classification(center_points,train_images, k):
# r[i][j]表示第i个样本是(1)否(0)属于第j类
r = np.zeros((train_images.shape[0],k)).astype(np.int16)
for i in range(train_images.shape[0]):
# 计算样本距离各个中心点的距离
distance = np.linalg.norm(center_points - train_images[i], axis=1)
# 将相距最近的中心点所属的类作为该样本的类
r[i][np.argwhere(distance==np.min(distance))[0][0]] = 1
return r
将每个样本成功归类后就要更新中心点的位置。 这里的中心点位置需要到该类中各个样本点的距离最短,因此是各个样本点的平均位置。同样,这里采用不同的距离度量方式也可以产生不同的结果。
```python
更新中心点位置
def updateCenterPoints(r, train_images, k): center_points = np.empty((k,train_images.shape[1])) for i in range(k): # 类内样本特征值之和除以样本数,即平均特征值 center_points[i] = np.sum(train_images*(np.repeat(r[:,i].reshape(1,-1),\ train_images.shape[1],axis=0).T),axis=0) / np.sum(r[:,i]) return center_points ```
2.1.3 聚类标签的获取
现在我们得到了十个聚类的中心点。经过训练,我们期望这些点能够对数据进行分类。然而我们不知道每个中心点对应的是哪一个标签。因为训练集中,每个类中有多个样本,可以让这些样本进行投票,每个样本为自身表示的标签进行投票,票数最多的标签就作为该中心点所代表的类的标签:
python
labels = (genLabels(r, train_labels, k)).astype(np.int16)
```python
对每个聚类打上标签
def genLabels(r, train_labels, k): labels = np.empty(10) # 第一层循环遍历每个聚类 for i in range(k): # 第二层循环统计该类中样本属于某一类的个数 max_count = 0 max_label = -1 # 分别对每个标签进行投票 for j in range(k): tmp = np.sum(train_labels[r[:,i]==1]==j) # 记录当前票数最多的标签与对应的票数 if tmp > max_count: max_count = tmp max_label = j labels[i] = max_label return labels ```
2.1.4 验证结果
在验证时,只需要比较验证样本与各个中心点的距离,选择距离最近的中心点所代表的类的标签作为预测结果,与实际的验证样本标签相比较即可。
python
right = 0
for i in range(test_images.shape[0]):
distance = np.linalg.norm(center_points - test_images[i], axis=1)
predict = labels[np.argwhere(distance==np.min(distance))[0][0]]
if predict == test_labels[i]:
right+=1
print(right,right/test_images.shape[0])
2.2 GMM
在GMM方法中,我们将不同的训练样本看做一个高斯混合模型的分布。不同的标签对应不同的分布,而每个分布由多个高斯分布组合而成。因此,对于每一类的分布都需要单独训练。
2.2.1 数据预处理
在GMM算法中,需要计算每个样本属于某个分布中的高斯分布的概率。而当特征向量的维数很高时,高斯分布的概率密度函数在特定点的大小就会特别的小。本次的实验中每个样本的维度为784,这么高的维度是不能够应用GMM的,因此首先要考虑对数据进行降维。这里我采用了
sklearn.decomposition
中的PCA模型,使用主成成分分析的方法对数据进行降维:
python
pca_model = PCA(n_components=50)
pca_model.fit(train_images.T)
train_images = pca_model.components_.T
pca_model.fit(test_images.T)
test_images = pca_model.components_.T
每个GMM应该用于拟合一类的样本,因此我们需要给样本进行分类。按照标签将训练样本分为十类,每一类单独用来训练一个GMM:
```python
将训练数据按照类别分组
type_num = 10 # 不同标签的个数 tmp = [[] for i in range(type_num)] for i in range(train_num): tmp[train_labels[i]].append(train_images[i])
train_images[i][j]表示类型为i的第j个样本
train_images = tmp[:][:]
```
2.2.2 训练过程
用每一类单独训练一个GMM:
```python
设置基本参数
k = 10 # 每个GMM中高斯模型的个数 t = 100 # 单个GMM迭代次数 GMM = [] # 记录每个类的GMM for i in range(type_num): GMM.append(genGMM(k,t,np.asarray(train_images[i]))) ```
训练单个GMM时,首先设置GMM的基本参数和初始值。需要混合高斯模型中,每个高斯分布的均值
gauss_means
、每个高斯分布的协方差矩阵
gauss_var
,以及这些高斯分布在模型中所占的比例
pi
。同时还需要样本属于某个分布的概率值
gamma
。分布的均值由数据集中随机抽取
k
个样本得到,协方差矩阵初始化为单位矩阵。比重和概率一开始都均等。
python
def genGMM(k,t,train_images):
# 每个高斯分布的均值,初始化为随机的k个样本点的位置
train_num = train_images.shape[0]
gauss_means = np.asarray(train_images[random.sample([i for i in range(train_num)],k)])
# 每个高斯分布的协方差矩阵
gauss_var = np.asarray([(1*np.eye(train_images.shape[1])) for j in range(k)])# 对角且相等
# gamma[i][j] 表示样本i属于第j个高斯分布的概率
gamma = np.ones((train_num, k)) / k
# 每个分布的比重
pi = gamma.sum(axis=0) / gamma.sum()
下面开始迭代更新参数。
python
for i in range(t):
本次实验采用EM算法来训练GMM。EM算法的执行过程简单来说,就是在给定的参数下得到对数似然函数的期望,然后进一步更新参数,使得原来的参数下得到的对数似然函数取得最大值。
E步骤的过程如下:
```python
E-step
计算对数似然函数
pdf = np.zeros((train_num, k)) for j in range(k): pdf[:, j] = pi[j] * multivariate_normal(gauss_means[j], gauss_var[j],allow_singular=True).pdf(train_images) log_likehood = np.mean(np.log(pdf.sum(axis=1))) print(log_likehood) ```
这里,我使用了
scipy
库的
multivariate_normal
来生成单个多元高斯分布模型。给定该分布的均值和协方差矩阵,可以得到其概率密度分布。调用
pdf
功能,给出样本,就可以得到该样本在该高斯分布下出现的概率。用
pdf[i][j]
表示在该高斯混合模型下,第
i
个样本属于第
j
个高斯分布的概率。
然后,进行M步骤,按照先前算出来的公式更新参数,使得当前的概率密度期望达到最大值:
```python
M-step
更新各个分布的参数
gamma = pdf / pdf.sum(axis=1).reshape(-1,1) pi = gamma.sum(axis=0) / gamma.sum() for j in range(k): gauss_means[j] = np.average(train_images, axis=0, weights=gamma[:, j]) cov = [np.dot((train_images[t]-gauss_means[j]).reshape(-1,1),(train_images[t]-gauss_means[j]).reshape(1,-1)) for t in range(train_num)] cov_sum = np.zeros((train_images.shape[1], train_images.shape[1])) for l in range(train_num): cov_sum += gamma[l][j] * cov[j] gauss_var[j] = cov_sum / np.sum(gamma[:,j]) ```
每个高斯混合模型需要保留各个高斯分布的均值、协方差,以及他们的权重,这三个参数决定了一个完整的高斯混合模型:
python
return [gauss_means, gauss_var, pi]
2.2.3 验证结果
在得到了所有的高斯混合模型后,需要测试时,将测试样本在每个高斯混合模型下算出其对数似然函数值,用于表示它符合各个高斯混合模型的概率。选择概率最高的那一个模型对应的标签作为预测结果:
python
right = 0
for l in range(test_images.shape[0]):
prob = []
# 依次使用每个GMM进行预测
for i in range(type_num):
pdf = np.zeros(k)
# 用每个模型中的每个高斯分布得到样本分布概率
for j in range(k):
pdf[j] = GMM[i][2][j] * multivariate_normal(GMM[i][0][j], GMM[i][1][j],allow_singular=True).pdf(test_images[l])
# 计算对数似然
prob.append(pdf.sum())
prob = np.asarray(prob)
# 选择对数似然最高的GMM对应的标签作为预测结果
ans = np.argwhere(prob==np.max(prob))[0][0]
if ans == test_labels[l]:
right+=1
print(right, right/test_images.shape[0])
三、实验结果与分析
3.1 K-means
使用MNIST数据集,同样进行100次迭代,K-means算法中,如果使用随机初始化中心点,准确率为58.9%;如果是基于距离选择的中心点,准确率为59.45%。二者差别不大。
K平均聚类的结果高度依赖于初始化的中心点。选择中心点的策略不同的话可能会得到不同的结果。我们希望选择的初始点有各个类别的代表性,能够很好地区分各个类别。这就要求选择的初始点相距较大。初始时如果是随机选择中心点,即随机选择数据样本作为初始中心,有可能选择了距离很近的多个样本作为初始中心从而导致结果不好。当然也有可能随机选择的中心点较优。总的来说,存在选择较差点的风险。为了避免选到太近的点作为中心点,可以使用基于距离的方法,即一开始随机选一个样本作为中心点,其他时候选择离各个中心最远的样本作为中心点。但是如果这样选择的话,问题在于可能选到异常值,导致聚类结果不准确。更好的方法是结合二者的思想,设定一个距离阈值,在所有超过距离阈值的点中随机选择,这样既避免了选择的点太近,又可以减少选到异常点的概率。
MNIST数据集为手写数字图片的集合。可以想见,相同的位置的像素点,不同的数字更有可能不同,而相同数字更有可能相同。K平均算法处理这样的数据是合理且有效的。
3.2 GMM
使用EM训练的GMM算法,初始化数据降维到50,每个GMM含有50个高斯分布,迭代10次,如果协方差矩阵初始化为对角且元素相等,准确率为37.86%;如果对角且不相等则为35.23%。下面的分析以协方差矩阵初始化为对角且元素相等的情况为例。
下图为第一个GMM训练时,对数似然函数值关于训练次数的变化:
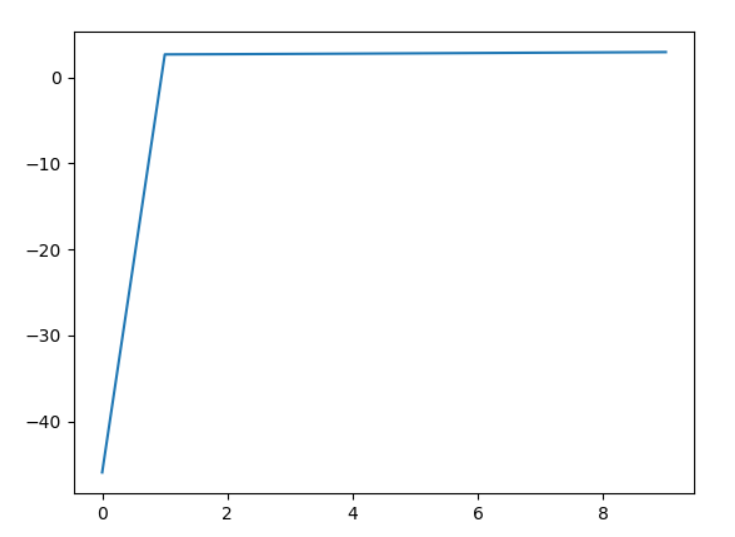
模型很快收敛了,但最终的对数似然函数的数值仍然较小,且几乎收敛,说明模型已经尽可能地拟合了数据,但拟合效果仍然不理想,模型不能很好地表示数据的分布。通过输出结果,我发现0、6等标签出现的概率高于其他标签,这说明这几个标签对应的GMM能更好地表示整个数据集,因此在这几个模型下会有更高的对数似然值,甚至比实际标签对应的模型算出来的对数似然值更高。这也侧面体现了模型拟合性能的不足。
可以看到,GMM算法的准确率非常低,而且实际训练过程中,训练时间相较K-means也更长。这应该是数据维度导致的。本次实验处理的数据维度高达784,而单个多变量高斯函数随着维度的上升,概率密度函数会越来越扁平。也就是说,在高维空间上,由高斯分布拟合的数据的概率会非常非常小,即便是在概率最大的均值处也会相当小。高达784维的数据根本不能简单地使用GMM算法运算。使用PCA方法降维又会产生样本信息的损失,且样本本身也不一定符合高斯分布,由此导致了结果性能较差。GMM更适合样本特征向量维度较小的情形。
总的来说,在MNIST数据集下,相比EM训练的GMM算法,K-means算法有着更好的性能。
参考文献
- 基于文本聚类的垂直搜索引擎系统设计与实现(北京工业大学·陈迪阳)
- 基于网络用户行为分析的用户推荐反馈系统的设计(北京化工大学·石钊)
- 基于聚类与加权矩阵分解的推荐算法研究(山东理工大学·郭蕊)
- 实时数据流自适应聚类及演化分析方法研究(苏州大学·夏月)
- 基于文本聚类的垂直搜索引擎系统设计与实现(北京工业大学·陈迪阳)
- 实时数据流自适应聚类及演化分析方法研究(苏州大学·夏月)
- 基于Web数据挖掘的推荐系统算法研究(河北工程大学·冯立娟)
- 推荐系统综合仿真平台评估框架的研究与实现(电子科技大学·施振兴)
- 基于密度的聚类算法研究及其在图书推荐系统中的应用(西北师范大学·黄梦莹)
- 基于网络爬虫的信息采集分类系统设计与实现(厦门大学·周茜)
- 基于三支聚类的推荐算法研究及应用(重庆邮电大学·康凯)
- 图结构数据聚类分析平台的研究及原型实现(东华大学·陈文俊)
- 基于三支聚类的推荐算法研究及应用(重庆邮电大学·康凯)
- 基于用户隐性行为的个性化推荐系统的设计及研究(南京财经大学·肖巧龙)
- 实时数据流自适应聚类及演化分析方法研究(苏州大学·夏月)
本文内容包括但不限于文字、数据、图表及超链接等)均来源于该信息及资料的相关主题。发布者:源码项目助手 ,原文地址:https://m.bishedaima.com/yuanma/36054.html